Diseñan un nuevo sistema para el ensamblaje de proteínas
20.12.2019
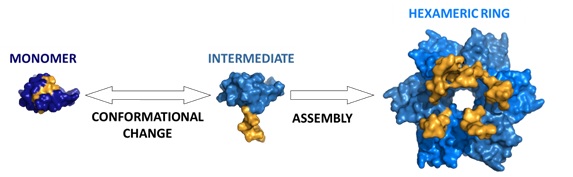
Diseño de un nuevo sistema de control alostérico de ensamblaje de proteínas mediante la creación y modulación racional de un interruptor molecular.
En la naturaleza existen numerosos ejemplos de macromoléculas biológicas con control alostérico, es decir, donde cambios en una región de la molécula afectan al comportamiento y función en otras regiones de la misma. Sin embargo, la reproducción de este tipo de comportamiento en moléculas sintéticas resulta muy complejo y hasta ahora los esfuerzos invertidos en este área solo han conseguido unos pocos resultados exitosos. De hecho, en el caso concreto de la creación de nuevos ensamblajes de diseño por ingeniería de proteínas, los resultados hasta ahora se han basado en un sistema tipo puzle, donde la superficie de cada una de las piezas rígidas que se van a ensamblar es modificada para producir una interacción estable, sin observarse cambios conformacionales. Ahora, investigadores de IMDEA Nanociencia y de la unidad asociada CNB-IMDEA, en colaboración con el Centro de Investigaciones Biológicas y las Universidades de Granada y California, han desarrollado un modelo que permite controlar de forma alostérica la formación y ensamblaje de proteínas mediante la inclusión de interruptores moleculares formados gracias a cambios en la estructura tridimensional de la proteína.
El trabajo, publicado en Nature Communications, demuestra el potencial de los interruptores alostéricos, utilizando como modelo el inhibidor de la quimotripsina 2 de la semilla de cebada. Esta proteína, inhibidor de proteasas que protege a la semilla de ser digerida, es una pequeña proteína monomérica que no oligomeriza de manera natural. Los investigadores, mediante técnicas de ingeniería de proteínas, han introducido cambios en la secuencia de aminoácidos que producen la formación de oligómeros (formando anillos simétricos de 6 y 12 subunidades) a través de un paso extra de control: un cambio conformacional que actúa como interruptor que permite controlar de manera externa cómo y cuándo la proteína oligomeriza. Luis Alberto Campos, primer autor del trabajo, explica que "este sistema modelo va a servir como base para obtener proteínas funcionales con control alostérico de la oligomerización, con posibles aplicaciones en ingeniería proteica, nanotecnología y biología sintética." El uso de estos sistemas de modulación de las transiciones entre monómero y oligómero aporta un mayor control, dinamismo y estabilidad en la formación de complejos macromoleculares y tiene aplicaciones en nuevos proyectos de investigación como la creación de anillos que interaccionen con otras moléculas, como metales o ácidos nucleicos, o el control en la formación de poros de membrana creados por péptidos antimicrobianos mediante proteínas de fusión.
Palabras clave: Computational biophysics, Molecular engineering, Nanobiotechnology, Protein folding, Structural biology
Más información:
Engineering protein assembies with allosteric control via monomer fold-switching. Campos LA, Sharma R, Alvira S, Ruiz FM, Ibarra-Molero B, Sadqi M, Alfonso C, Rivas G, Sanchez-Ruiz JM, Romero-Garrido A, Valpuesta JM, Muñoz V. Nat Commun 2019 Dec 13;10(1):5703. doi: 10.1038/s41467-019-13686-1
Contacto
IMDEA Nanociencia - Oficina de Divulgación
Esta dirección de correo electrónico está siendo protegida contra los robots de spam. Necesita tener JavaScript habilitado para poder verlo.
+34 912 998712
Twitter: @imdea_nano
Facebook: @imdeananociencia
Fuente: CNB, IMDEA Nanociencia, Unidad Asociada de Nanobiotecnología IMDEA Nanociencia-CNB, Madrid, Spain




