Los factores de transcripción eucariotas pueden rastrear y controlar sus genes diana usando antenas de ADN
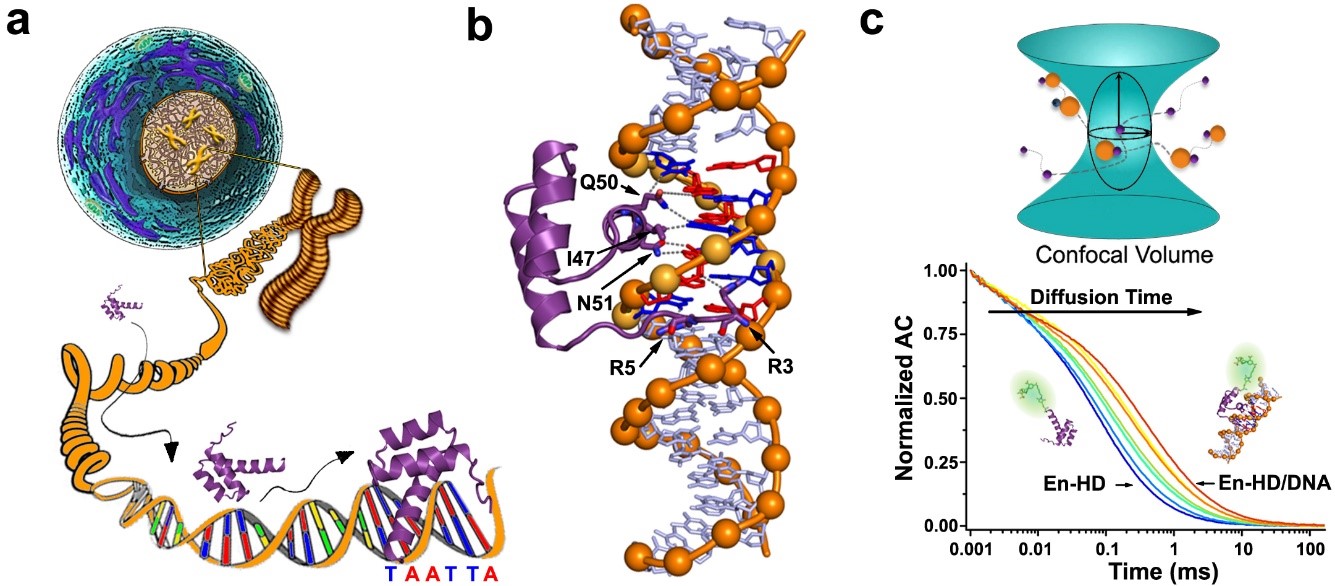
El homeodominio del factor de transcripción Engrailed (EngHD) uniéndose a su sitio específico en el DNA. a) Representación pictórica de los desafíos que implica la localización del sitio de unión específico en el ADN genómico de una célula eucariota. b) Estructura 3D del complejo específico entre EngHD y el ADN (PDB: 1HDD). c) Diagrama esquemático de cómo determinar la unión de EngHD al ADN usando la técnica de FCS.
05.02.2020
Los factores de transcripción eucariotas (FT) funcionan uniéndose a sitios de reconocimiento en el ADN de 6-10 pb ubicados cerca de sus genes diana, que se encuentran dispersos a lo largo de extensos genomas. Ese proceso ha de superar enormes desafíos de especificidad, eficiencia y celeridad a través de un mecanismo molecular que sigue siendo aún poco conocido. Combinando experimentos biofísicos, teoría y análisis bioinformáticos, este trabajo disecciona la interacción entre el dominio de unión al ADN de Engrailed, un FT de Drosophila, y las regiones reguladoras de sus genes diana. La afinidad de unión se amplifica enormemente por las regiones de ADN que flanquean el sitio de reconocimiento, y que contienen largas extensiones de repeticiones degeneradas del sitio diana. Tal organización del ADN opera como una antena que atrae moléculas del FT en un intercambio promiscuo entre miríadas de sitios de unión de afinidad intermedia. La antena asegura un suministro local de FT, permite el rastreo del gen, así como un control preciso de la ocupación basal del sitio diana. Este mecanismo arroja luz sobre datos de expresión génica no descifrados aún y sugiere nuevas estrategias de ingeniería para controlar la expresión génica.
Un equipo de investigación de IMDEA NANOCIENCIA, liderado por el Prof. Víctor Muñoz (actualmente en UCMerced: https://www.ucmerced.edu/), ha abordado una cuestión fundamental en la regulación de genes eucariotas, utilizando métodos biofísicos para diseccionar el proceso, modelos de mecánica estadística para integrar y racionalizar los resultados, y análisis bioinformático para explorar aún más las implicaciones funcionales. Los FT eucariotas rastrean sus genes diana, controlan la ocupación del sitio de unión específico y coordinan su unión con la de otras moléculas para formar el complejo de transcripción. Estos procesos han de involucrar modos de interacción con el ADN que van más allá de la unión inespecífica y la difusión facilitada 1D y / o 2D. Al centrarse en las regiones de ADN que flanquean el sitio diana de genes reales, han descubierto y caracterizado un mecanismo molecular que permite tales funciones. El mecanismo explota la tendencia natural de las biomoléculas a exhibir frustración energética, en este caso manifiesta por la promiscuidad de unión. Particularmente, ven que la afinidad del FT de Drosophila por las regiones reguladoras de sus genes diana está fuertemente amplificada por largas extensiones de repeticiones degeneradas de la secuencia consenso, presentes en dichas regiones. La combinación de un FT promiscuo y una región de ADN rica en estas repeticiones degeneradas funciona como una antena de transcripción. Este trabajo aporta un mecanismo elegante que arroja nueva luz sobre cómo operan los FT eucariotas a nivel molecular y explica varias paradojas de los datos de expresión de genes eucariotas existentes. Estos dispositivos moleculares proporcionan un nivel adicional de control transcripcional eucariota en el que el tamaño y el perfil de secuencia de la antena pueden ser diseñados, ya sea por la evolución o por los científicos, para modular la ocupación del sitio, la rapidez de respuesta y los niveles de expresión génica.
Este trabajo ha sido financiado por el Consejo Europeo de Investigación a través de la subvención ERC-2012-ADG-323059. M.C. reconoce el apoyo de una beca Juan de La Cierva del Ministerio de Economía y Competitividad de España. V.M. reconoce el apoyo de W.M. Keck Foundation y National Science Foundation a través de las subvenciones NSFCREST-1547848 (Centro de Máquinas Celulares y Biomoleculares) y NSF-MCB1616759.
Palabras clave: Factores de transcripción, Regulación de la expresión génica en Eucariotas, Biofísica de interacciones ADN-Proteína, FCS, Engrailed.
Reference:
M. Castellanos et al. Nat. Comm. (2020)
https://doi.org/10.1038/s41467-019-14217-8
Contacto:
Dr. Milagros Castellanos
Esta dirección de correo electrónico está siendo protegida contra los robots de spam. Necesita tener JavaScript habilitado para poder verlo.
Prof. Víctor Muñoz
Esta dirección de correo electrónico está siendo protegida contra los robots de spam. Necesita tener JavaScript habilitado para poder verlo.
IMDEA Nanociencia - Oficina de Divulgación
Esta dirección de correo electrónico está siendo protegida contra los robots de spam. Necesita tener JavaScript habilitado para poder verlo.
+34 912998712




